Berlin – Wissenschaftler*innen des Berlin Institute of Health in der Charité (BIH) und des MDC haben gemeinsam mit Kolleg*innen vom Heidelberger Institut für Stammzelltechnologien und Experimentelle Medizin (HI-STEM) am Deutschen Krebsforschungszentrum (DKFZ) sowie weiteren Forscher*innen aus Heidelberg und Barcelona die Entwicklung von Blutzellen detailliert nachverfolgt: Sie kombinierten Methoden, mit denen man die Aktivität der Gene innerhalb der Zelle analysiert mit dem Nachweis von Proteinmolekülen auf der Oberfläche der Zelle. Sie waren so in der Lage, die Informationen tausender einzelner Zellen aus Blut und Knochenmark gleichzeitig zu erfassen und damit die unterschiedlichen Entwicklungsstadien der verschiedenen Zelltypen eindeutig zu erkennen. Ihre Ergebnisse, die wichtig für die Diagnose und Therapie von Blutkrebserkrankungen sind, haben die Wissenschaftler*innen nun in der Zeitschrift Nature Immunology veröffentlicht.
Der Laie spricht bei Blutzellen von roten und weißen Blutkörperchen und Blutplättchen. Für Experten ist die Unterscheidung sehr viel detaillierter, vor allem bei den weißen Blutkörperchen, den Immunzellen, existieren viele verschiedene Zelltypen. Allen gemeinsam ist der Ursprung aus Blutstammzellen im Knochenmark. Wenn diese sich teilen und in verschiedene Zellen differenzieren, entsteht die Vielfalt der Blutzellen. „Bislang haben wir die verschiedenen Zellen anhand der Proteine auf ihrer Oberfläche voneinander unterschieden“, erklärt Ko-Letztautor der aktuellen Arbeit Dr. Simon Haas, Leiter der Nachwuchsgruppe Blutkrebs, Stammzellen und Präzisionsmedizin am BIH, die am Berliner Institut für molekulare Systembiologie (BIMSB) des Max-Delbrück-Centrums für Molekulare Medizin in der Helmholtz-Gemeinschaft angesiedelt ist. „Dazu nutzen wir die FACS-Analyse, oder Durchflusszytometrie, bei der Zellen mit der Hilfe von Antikörpern gegen rund 20 Oberflächenmoleküle analysiert werden“, berichtet Haas. „Das geht schnell und ist kostengünstig, ist aber nicht so genau.“
Mittlerweile gibt es sehr viel genauere Methoden, um Zellen zu untersuchen. Mit der Single-Cell-Analyse können Biowissenschaftler die verschiedenen messenger RNA (mRNA)-Moleküle in einzelnen Zellen bestimmen. Diese Botenstoffe überbringen die Information aus dem Erbgut im Zellkern, der DNA, in das Zellplasma, wo die mRNA in Eiweiß (Protein) übersetzt wird. In jeder Zelle ist der Gehalt und die Zusammensetzung der mRNA individuell verschieden. „Das ermöglicht uns, viel feinere Unterschiede zwischen Zellen festzustellen“, sagt Simon Haas. „So können wir mittlerweile verschiedene Entwicklungsstadien der Zellen erkennen, was zum Beispiel wichtig ist, um Blutkrebs genauer zu diagnostizieren oder den Alterungsprozess von Blutzellen zu verstehen.“ Für die tägliche Routine im Klinikbetrieb ist die Single Cell Diagnostik jedoch noch viel zu aufwändig und teuer. „Wir haben uns deshalb überlegt, ob man die Single-Cell-Analyse nicht mit der FACS-Analyse kombinieren kann.“
Hochpräzise Karte der Blutentwicklung
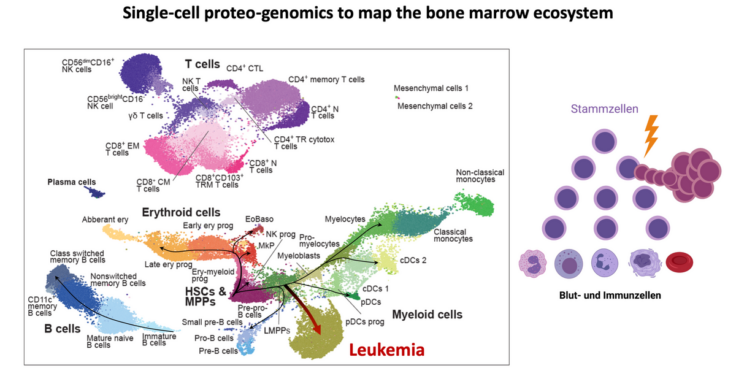
Dazu untersuchten die Wissenschaftler*innen um Simon Haas gemeinsam mit ihren Kolleg*innen am Heidelberger Institut für Stammzelltechnologien und Experimentelle Medizin (HI-STEM) am Deutschen Krebsforschungszentrum (DKFZ), am Europäischen Molekularbiologischen Labor (EMBL) in Heidelberg und am Centre for Genomic Regulation (CRG) in Barcelona zunächst hunderttausende von Blutzellen verschiedener Stadien im Labor mit der Single-Cell-Analyse und erstellten so eine hochpräzise Karte der Entwicklung von Zellen des Blutsystems, sowohl von gesunden Spendern als auch von Leukämiepatienten. So konnten sie die normalen Alterungsprozesse nachvollziehen, aber auch erkennen, wie aus Stammzellen Leukämiezellen entstehen. Dabei entdeckten sie auch neue Markerproteine auf den Zelloberflächen, die wesentlich genauere Unterscheidungen zwischen den verschiedenen Zelltypen erlaubten.
„Das ist ganz wichtig, zum Beispiel bei der Diagnose und Therapie von Blutkrebserkrankungen“, betont Simon Haas. „Hier entwickeln sich die Zellen nicht in funktionstüchtige fertige Blutzellen, sondern bleiben während ihrer Entwicklung auf einer bestimmten Stufe stehen und vermehren sich immer weiter. Das geht so lange, bis das Blut überschwemmt ist von weißen Blutkörperchen, das Merkmal der Leukämie, des „weißen Blutes“. Damit wir diese Zellen zielgerichtet bekämpfen können, ist es notwendig, sie genau zu kennen und von gesunden Blutzellen unterscheiden zu können.“
Kombinierte Analyseverfahren für präzise Diagnostik
Die Wissenschaftler verknüpften daher die neu gefundenen Informationen über die verschiedenen Zellstadien mit den herkömmlichen Oberflächenmarkern, die beim FACS zum Einsatz kommen. Ergänzt um die neuen Markerproteine konnten sie mithilfe künstlicher Intelligenz die Ergebnisse aus den Single-Cell-Analysen mit den FACS-Ergebnissen kombinieren. „Wir benutzen jetzt neue Kombinationen von Antikörpern, die uns die KI vorschlägt, um ganz bestimmte Zellstadien besser identifizieren zu können“, erklärt Haas.
Die Nachwuchsgruppe von Simon Haas gehört dem gemeinsamen Forschungsfokus „Single-Cell-Ansätze für die personalisierte Medizin“ an, den das BIH in der Charité gemeinsam mit dem MDC und der Charité – Universitätsmedizin Berlin gegründet hat. Die klinische Anwendung seiner Ergebnisse verfolgt Simon Haas nun mit seinen klinischen Partnern an der Charité, den Direktoren der Medizinischen Kliniken mit Schwerpunkt Hämatologie, Onkologie und Tumorimmunologie, Professor Lars Bullinger am Charité Campus Virchow-Klinikum (CVK) sowie Professor Ulrich Keller am Charité Campus Benjamin Franklin (CBF). „Unser Ziel ist, Leukämien so besser zu diagnostizieren und die Blutkrebszellen präzise bekämpfen zu können.“
Veröffentlichung: Nature Immunology „Single-cell proteo-genomic reference maps of the hematopoietic system enable the purification and massive profiling of precisely defined cell states“; Sergio H. Triana, Dominik Vonficht, Lea Jopp-Saile, Simon Raffel, Raphael Lutz, Daniel Leonce, Magdalena Antes, Pablo Hernández-Malmierca, Diana Ordoñez-Rueda, Beáta Ramasz, Tobias Boch, Johann-Christoph Jann, Daniel Nowak, Wolf-Karsten Hofmann, Carsten Müller-Tidow, Daniel Hübschmann, Theodore Alexandrov, Vladimir Benes, Andreas Trumpp, Malte Paulsen, Lars Velten, Simon Haas.